商机详情 -
内蒙古RNA蛋白互作检测RIP Sequence
RIP-qPCR实验(RNA Immunoprecipitation followed by quantitative PCR)是一种用于研究细胞内特定蛋白质与RNA相互作用的技术。该技术结合了免疫沉淀(Immunoprecipitation)和实时荧光定量PCR(quantitative PCR,qPCR)的方法,旨在识别和定量与特定蛋白质结合的RNA分子。在RIP-qPCR实验中,首先使用针对目标蛋白质的特异性抗体进行免疫沉淀,将与该抗体结合的蛋白质-RNA复合物从细胞裂解液中分离出来。随后,通过洗涤步骤去除非特异性结合的分子,保留与目标蛋白质特异性结合的RNA。接下来,从免疫沉淀复合物中提取RNA,并将其逆转录为cDNA。然后,利用特异性引物进行qPCR反应,以定量检测与目标蛋白质结合的特定RNA分子的丰度。通过比较不同样品中目标RNA的相对表达水平,可以评估蛋白质与RNA之间的结合强度和特异性。RIP-qPCR实验在生物学研究中具有广泛应用,可用于研究转录后调控、RNA转运、RNA稳定性以及非编码RNA与蛋白质相互作用等方面的问题。该技术为揭示细胞内基因表达调控的复杂网络提供了有力工具。RIP-seq实验的研究对象主要包括细胞内与特定蛋白质结合的RNA分子。内蒙古RNA蛋白互作检测RIP Sequence
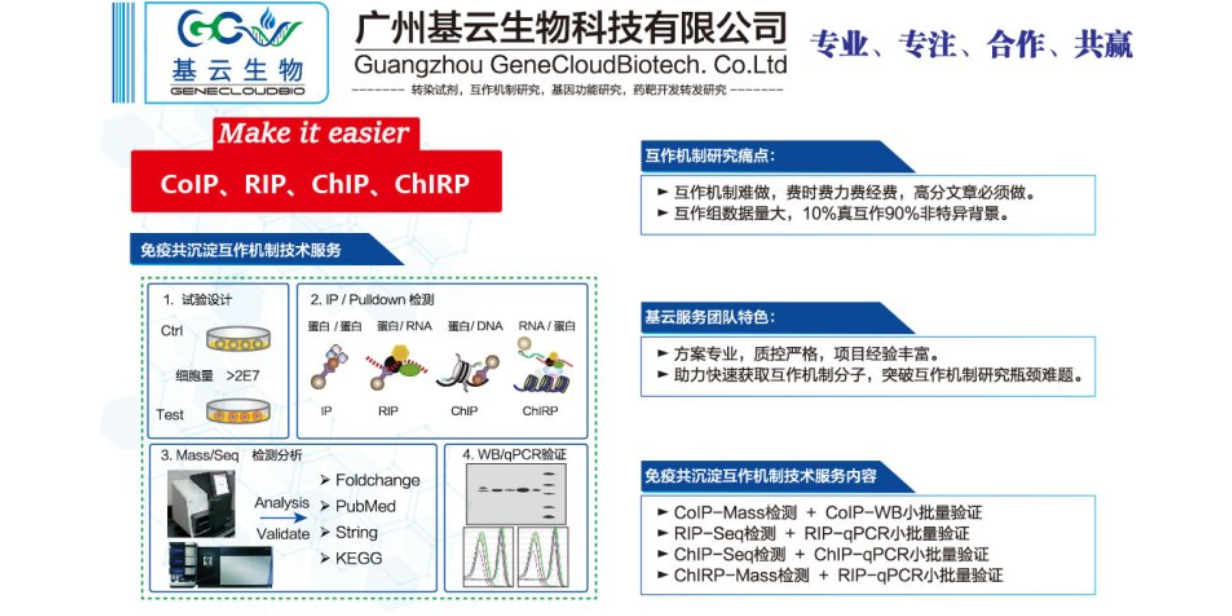
RIP-qPCR(RNA免疫沉淀结合实时荧光定量PCR)是一种用于研究RNA与蛋白质相互作用的技术。以下是其基本实验路线:细胞准备:首先,收集目标细胞或组织,并进行适当的细胞裂解,以获得包含RNA-蛋白质复合物的裂解液。在此过程中,需要添加RNase抑制剂,以保护RNA不被降解。抗体结合:将特异性抗体添加到细胞裂解液中,这些抗体能够与目标蛋白质(即与RNA结合的蛋白质)特异性结合。通过免疫反应,抗体与目标蛋白质形成复合物。免疫共沉淀:加入蛋白A/G磁珠或类似的亲和树脂,这些磁珠能够与抗体-目标蛋白质复合物结合。然后,通过磁力将复合物沉淀下来,同时去除非特异性结合的蛋白质。洗涤:使用适当的洗涤缓冲液多次洗涤磁珠,以去除非特异性结合的蛋白质和其他污染物,确保结果的准确性。RNA提取与反转录:从沉淀的复合物中提取RNA,并在此过程中再次添加RNase抑制剂以保护RNA。随后,使用逆转录酶将提取的RNA反转录为cDNA。qPCR检测:后续通过实时荧光定量PCR(qPCR)技术检测特定RNA序列的含量,从而验证RNA与目标蛋白质的相互作用。这就是RIP-qPCR的基本实验路线,它提供了一种有效的方法来研究细胞内RNA与蛋白质的相互作用。安徽RNA免疫共沉淀RIP RT-PCRRIP-seq主要应用于识别和分析与特定RNA结合蛋白(RBP)结合的RNA分子。
RIP-seq(RNA Immunoprecipitation sequencing)是一种用于研究细胞内RNA与蛋白质结合情况的高通量测序技术。其基本原理是通过目标蛋白的抗体将相应的RNA-蛋白质复合物沉淀下来,然后经过分离纯化,对结合在复合物上的RNA进行高通量测序分析。该技术利用抗体或表位标记物捕获细胞核内或细胞质中的内源性RNA结合蛋白,从而避免非特异性的RNA结合。通过免疫沉淀,RNA结合蛋白及其结合的RNA被一起分离出来。之后,结合的RNA序列通过高通量测序方法进行鉴定和分析。RIP-seq技术结合了免疫沉淀和高通量测序技术,具有高通量、高分辨率和高灵敏度的特点,能够详细、准确地揭示细胞内RNA与蛋白质的相互作用网络。该技术不仅可以用于发现新的RNA结合蛋白和它们的靶标RNA,还可以用于研究RNA在转录后调控、细胞代谢、疾病发生、发展等过程中的功能和机制。总之,RIP-seq技术是一种强大的工具,为研究细胞内RNA与蛋白质的相互作用提供了有力的手段,有助于深入了解细胞内基因表达调控的复杂网络。
RIP-qPCR实验技术具有多个优点和一些潜在的缺点。优点:特异性高:RIP-qPCR结合了免疫沉淀和qPCR技术,能够特异性地识别并结合目标RNA结合蛋白(RBP)及其结合的RNA,降低非特异性结合的可能性。灵敏度高:qPCR技术具有高灵敏度,能够检测到低丰度的RNA分子,使得RIP-qPCR能够准确测量细胞中RNA与蛋白质的相互作用。定量准确:通过实时监测荧光信号,RIP-qPCR可以对目标RNA进行精确定量,提供可靠的定量数据。应用范围大:RIP-qPCR技术适用于多种生物样本和实验条件,可用于研究不同细胞类型、组织或生物体中的RNA-蛋白质相互作用。缺点:技术复杂性:RIP-qPCR涉及多个步骤,包括细胞裂解、免疫沉淀、RNA提取、逆转录和qPCR等,操作相对复杂,需要经验丰富的实验人员。抗体依赖性:实验结果的准确性和特异性高度依赖于所使用的抗体的质量和特异性。非特异性抗体可能导致假阳性或假阴性结果。RNA易降解:RNA分子在操作过程中容易降解,特别是在不适当的实验条件下,如存在RNase污染或操作时间过长。综上所述,RIP-qPCR实验技术具有高特异性和灵敏度,能够准确测量RNA与蛋白质的相互作用,但操作复杂、抗体依赖性强、RNA易降解以及成本较高是其潜在的缺点。RIP-qPCR实验的基本实验步骤主要包括哪些。

RIP-seq和RIP-qPCR实验都是研究RNA与蛋白质相互作用的实验方法,但存在一些异同点。相同点:两者都基于RNA免疫沉淀(RIP)技术,利用特定蛋白的抗体将RNA-蛋白质复合物沉淀下来,以研究RNA与蛋白质的相互作用。两者都需要对实验条件进行优化,以确保实验的特异性和准确性。不同点:实验目的:RIP-seq主要用于筛选与目标蛋白结合的未知RNA,绘制全基因组范围的RNA与蛋白质相互作用图谱,而RIP-qPCR则用于验证与目标蛋白结合的已知RNA。数据分析:RIP-seq产生高通量测序数据,需要生物信息学分析以识别与蛋白质结合的RNA序列;而RIP-qPCR产生定量PCR数据,通过相对定量方法分析特定RNA与蛋白质的结合情况。应用范围:RIP-seq更适合于发现新的RNA与蛋白质的相互作用,并研究其在全基因组范围内的分布和特征;而RIP-qPCR更适用于特定RNA与蛋白质相互作用的验证和定量研究。总之,RIP-seq和RIP-qPCR实验在研究RNA与蛋白质的相互作用时各有优势,研究者可根据具体需求选择合适的方法。对于科研新手来说,在进行RIP-qPCR实验时,需要特别注意哪些问题。RNA蛋白相互作用RIP Seq检测
RIP-seq和RIP-qPCR实验有哪些异同点。内蒙古RNA蛋白互作检测RIP Sequence
RIP-qPCR实验技术的原理是基于RNA免疫沉淀(RNA Immunoprecipitation, RIP)与实时荧光定量PCR(quantitative real-time PCR, qPCR)的结合。首先,通过RIP技术,利用抗体特异性地识别并结合目标RNA结合蛋白(RBP),将RBP与其结合的RNA一起沉淀下来。这一步骤依赖于抗体与RBP之间的特异性相互作用,确保只有与目标RBP结合的RNA被沉淀。接下来,从沉淀的复合物中提取RNA,并通过逆转录将其转化为cDNA。然后,利用qPCR技术对特定的RNA分子进行定量检测。在qPCR反应中,通过荧光信号的实时监测,可以准确测量PCR产物的累积量,从而实现对目标RNA的定量分析。综上所述,RIP-qPCR实验技术的原理是通过特异性抗体沉淀目标RBP及其结合的RNA,然后利用qPCR对沉淀下来的RNA进行定量检测。这项技术结合了RIP的特异性和qPCR的灵敏性,为研究细胞内RNA与蛋白质的相互作用提供了有力工具。通过这种方法,可以深入了解RNA与蛋白质在细胞内的结合情况,揭示转录后调控网络的动态过程。内蒙古RNA蛋白互作检测RIP Sequence