商机详情 -
染色体免疫沉淀ChIP RT-PCR
CHIP实验需要注意点就是抗体的性质。抗体不同和抗原结合能力也不同,免染能结合未必能用在IP反应。建议仔细检查抗体的说明书。特别是多抗的特异性是问题。其次,要注意溶解抗原的缓冲液的性质。多数的抗原是细胞构成的蛋白,特别是骨架蛋白,缓冲液必须要使其溶解。为此,必须使用含有强界面活性剂的缓冲液,尽管它有可能影响一部分抗原抗体的结合。另一面,如用弱界面活性剂溶解细胞,就不能充分溶解细胞蛋白。即便溶解也产生与其它的蛋白结合的结果,抗原决定族被封闭,影响与抗体的结合,即使IP成功,也是很多蛋白与抗体共沉的悲惨结果。再次,为防止蛋白的分解,修饰,溶解抗原的缓冲液必须加蛋白每抑制剂,低温下进行实验。每次实验之前,首先考虑抗体/缓冲液的比例。抗体过少就不能检出抗原,过多则就不能沉降在beads上,残存在上清。缓冲剂太少则不能溶解抗原,过多则抗原被稀释。ChIP-qPCR和ChIP-seq在实验流程、分辨率和应用范围上存在异同点,应根据具体需求选择合适的技术方法。染色体免疫沉淀ChIP RT-PCR
Q:ChIP-Seq和ChIP-qPCR有何异同?A:染色质免疫共沉淀(ChIP)所获得的DNA产物,在ChIP-Seq中通过高通量测序的方法,在全基因组范围内寻找目的蛋白(转录因子、修饰组蛋白)的DNA结合位点片段信息;ChIP-qPCR需要预设待测的目的序列,针对目的序列设计引物,以验证该序列是否同实验蛋白结合互作。
Q:染色质片段大小在哪个范围比较合适?A:对于ChIP-seq,片段在200-500bp左右是合适范围;对于ChIP-qPCR,片段在200-800bp左右适宜。
Q:植物样本处理和动物组织/细胞有何区别?A:植物组织由于细胞壁、气腔等结构的存在,会给交联缓冲液的作用带来困难,因此相对于动物组织/细胞来说,往往需要在抽真空条件下进行交联,而该步奏是一个需要经验及优化的过程。
Q:ChIP-Seq中的测序DNA样本需要多少产量?A:通常是≥10ng。
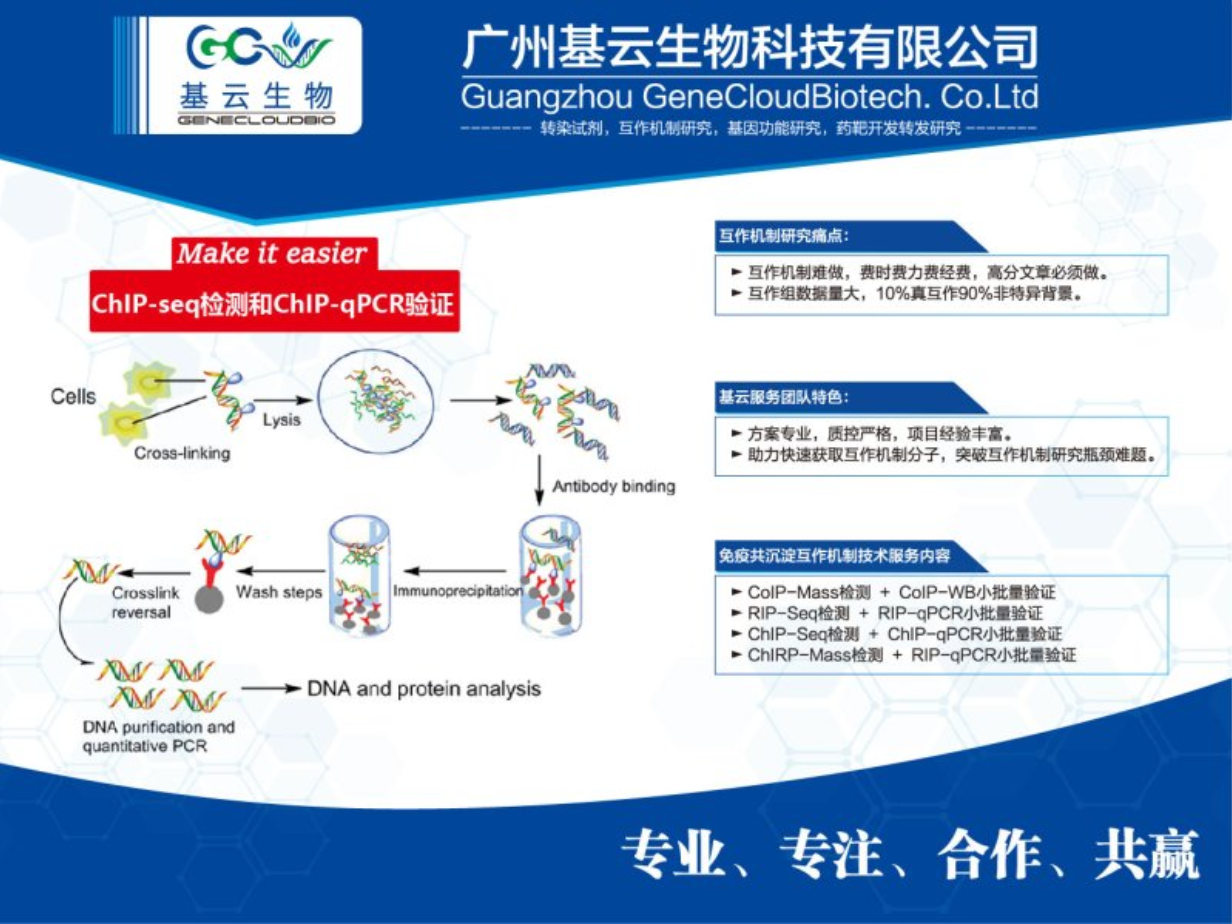
Q:ChIP风险如果判断A:ChIP实验以标签来判断实验风险,重组标签的转录因子>内源转录因子>组蛋白;当以重组蛋白作为靶蛋白时,重组蛋白同内源蛋白可能存在结合活性、结合位点差异;以标签抗体进行ChIP时、染色质结合位点本身会被内源蛋白竞争,这些都会影响到ChIP过程的特异性捕获效率。 chromatin免疫共沉淀检测ChIP PCRChIP-qPCR实验流程包括交联细胞、裂解细胞核、切割染色质、免疫沉淀、洗涤、反交联、DNA纯化和QPCR反应等。

ChIP不仅可以检测体内反式因子与DNA的动态作用,还可以用来研究组蛋白的各种共价修饰与基因表达的关系。近年来,这种技术得到不断的发展和完善。采用结合微阵列技术在染色体基因表达调控区域检查染色体活性,是深入分析AI症、心血管疾病以及神经系统紊乱等疾病的主要代谢通路的一种非常有效的工具。它的原理是在保持组蛋白和DNA联合的同时,通过运用对应于一个特定组蛋白标记的生物抗体,染色质被切成很小的片断,并沉淀下来。IP是利用抗原蛋白质和抗体的特异性结合以及细菌蛋白质的“proreinA”特异性地结合到免疫球蛋白的FC片段的现象活用开发出来的方法。目前多用精制的proreinA预先结合固化在argarose的beads上,使之与含有抗原的溶液及抗体反应后,beads上的proreinA就能吸附抗原达到精制的目的。
ChIP-qPCR实验虽然是一种有效的研究蛋白质与DNA相互作用的方法,但也存在一些缺点。首先,ChIP-qPCR实验通常只能针对已知基因或基因区域进行分析,无法在全基因组范围内寻找未知的结合位点,这在一定程度上限制了其应用范围。其次,该实验方法的分辨率相对较低,可能无法精确到具体的结合位点,只能确定大致的结合区域。这可能会影响对转录因子等蛋白质在基因调控中具体作用机制的深入理解。此外,ChIP-qPCR实验的结果可能受到多种因素的影响,如抗体的特异性、交联条件、染色质片段化效果等。这些因素可能导致实验结果的稳定性和可重复性受到一定程度的影响。另外,ChIP-qPCR实验需要相对较多的起始材料,且实验步骤较为繁琐,需要经验丰富的实验人员进行操作。这可能会增加实验的难度和成本,限制其在一些实验室的广泛应用。综上所述,尽管ChIP-qPCR实验在研究蛋白质与DNA相互作用方面具有一定的应用价值,但也存在一些缺点需要在实际应用中予以注意和克服。ChIP-qPCR实验虽然是一种有效的研究蛋白质与DNA相互作用的方法,但也存在一些缺点。
使用ChIP-seq快速确定下游靶标涉及多个关键步骤:首先,进行ChIP实验以富集与目标蛋白(如转录因子)结合的DNA片段。在这一步中,确保使用高质量的抗体以特异性地捕获目标蛋白与DNA的复合物。接着,将富集的DNA片段进行高通量测序。测序产生的数据将提供全基因组范围内目标蛋白的结合位点信息。然后,对测序数据进行生物信息学分析。这包括将测序读段比对到参考基因组上,识别并注释峰值区域,这些峰值区域表示目标蛋白与DNA的潜在结合位点。接下来,分析峰值区域在基因组中的分布,以确定下游靶标。特别关注那些位于基因启动子、增强子等调控区域的峰值,因为这些区域通常与基因表达调控密切相关。此外,还可以整合其他组学数据(如转录组学、表观遗传学数据等),以进一步验证和解释目标蛋白与下游靶标之间的调控关系。另外,通过实验验证(如qPCR、基因敲除或过表达等)来确认下游靶标的功能和调控作用。综上所述,通过ChIP-seq实验结合生物信息学分析和实验验证,可以快速而准确地确定下游靶标,并揭示目标蛋白在基因表达调控网络中的作用机制。染色质免疫沉淀(ChIP)实验注意事项有哪些。海南染色质蛋白相互作用检测ChIP
ChIP-seq(染色质免疫沉淀测序)是一种强大的实验技术,广泛应用于多个生物学领域。染色体免疫沉淀ChIP RT-PCR
ChIP-Seq检测原理:ChIP-Seq检测原理和RIP-Seq类似,不同的是前者利用目的蛋白抗体将相应的DNA-蛋白复合物沉淀下来,然后分离纯化捕获DNA,结合高通量测序技术对目标DNA进行测序分析。ChIP-Seq服务要点和RIP-Seq类似,精简如下:(1)试验设计:同RIP-Seq。(2)蛋白表达和细胞量:比RIP-Seq细胞用量要求大,建议不少于10e7(金标准:320g离心沉淀100ul)。(3)抗体关键质控:同IP-Mass和RIP-Seq。(4)IP送样建议:细胞培养好后,收集前,先进行交联,再收样冻存。(5)互作DNA筛选和验证:同RIP-Seq。ChIP-Seq优劣势:优势:高通量获得目的蛋白的专属DNA互作库。劣势:技术门槛高,一般需要整包交给专业的服务商开展检测。ChIP-Seq应用扩展:(1)蛋白DNA相互作用数据,是探究转录调控机制研究的重要内容,体现机制研究的深度,能显著提高临床基础类研究文章的档次。(2)蛋白DNA互作组检测,常用于蛋白的转录调控研究,如转录因子,转录调控蛋白等。(3)蛋白DNA互作,其结合DNA的区域,是进一步研究互作机制和功能的关键内容,能够显著提高机制研究的高度。染色体免疫沉淀ChIP RT-PCR